INTRODUCCIÓN
La tuberculosis constituye una enfermedad de alto riesgo que afecta al sistema respiratorio y causa alrededor de 1.5 millones de muertes. África y Asia concentran la mayor proporción de casos confirmados, mientras que los países como América Latina y el Caribe han registrado un aumento en muertes por esta enfermedad. La tasa promedio de detección es relativamente alta, no obstante, se establece que más del 50% de casos que no se detectan de forma temprana, lo que refleja las desigualdades en la capacidad de control y diagnóstico oportuno de la enfermedad. Por otro lado, los casos de tuberculosis se incrementaron un 19% en América Latina, contrastando la disminución del 8,7% a nivel mundial (Liu et al., 2024). Esta problemática es causada por los recursos limitados y que afecta directamente a la tasa de mortalidad y control sobre la propagación de la enfermedad (Sánchez Figueroa et al., 2024).
Estudios han corroborado que enfoques basados en CNN para la detección de enfermedades con un amplio conjunto de datos radiográficos, pueden alcanzar altos niveles de efectividad, especialmente cuando se emplean arquitecturas de CNN preentrenadas como: ResNet, VGG, Inception, DenseNet. Arquitecturas que al ser combinadas con segmentación pulmonar mediante U-Net, obtienen una precisión del 98,6% y un área bajo la curva (AUC) cercano a 1,0 (Rahman et al., 2020).
Otros autores explican el uso de modelos como AlexNet, InceptionV3 y DenseNet permiten clasificar estados en objetos mediante el uso de imágenes que facilitan la toma de decisiones precisas, mejorando la precisión diagnóstica (Romero García et al., 2025). Así mismo, se ha explorado el desarrollo de modelos híbridos combinando transformers con ResNet, que logra aprovechar las capacidades de vision transformers en la captura de relaciones entre imágenes, enfoque que mejora la precisión en el diagnóstico (Kotei y Thirunavukarasu, 2024). Por otro lado, se ha desarrollado una red neural gráfica bajo el nombre de TBNet, diseñada para detectar tuberculosis a partir de imágenes tomográficas computarizadas que se distingue por su capacidad de captura en relaciones espaciales entre regiones en la anatomía, detectando lesiones asociadas a tuberculosis y una alta confiabilidad en el diagnóstico automatizado (Lu et al., 2022).
El objetivo de este estudio fue realizar una comparación a diferentes modelos de CNN que apoyen al diagnóstico de tuberculosis de forma más precisa y objetiva, como un método complementario de diagnóstico para esta enfermedad.
MÉTODOS
Este estudio adoptó la metodología CRISP-DM, reconocida como el modelo estándar para la implementación de proyectos de minería de datos. Su estructura se compone de seis fases: comprensión del negocio, comprensión de los datos, preparación de los datos, modelado, evaluación y despliegue. Las fases se relacionan y permiten una aproximación iterativa y flexible al análisis de datos, adaptándose a las necesidades específicas de cada proyecto (Schröer et al., 2021). Su aplicabilidad en contextos de salud ha sido validada en investigaciones recientes, donde ha guiado con éxito el desarrollo de soluciones basadas en Machine Learning para la detección de enfermedades (Tamayo y Sauñe, 2024). El seguimiento de la metodología CRISP-DM para el entrenamiento de los modelos, se ha diseñado siguiendo los fundamentos de la figura 1.
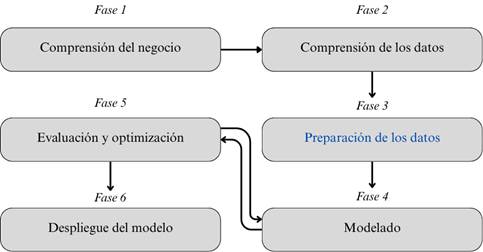
Figura 1. Metodología CRISP-DM.
Elaboración: Los autores.
La elección de un enfoque experimental basado en Deep Learning para el diagnóstico de tuberculosis en radiografías de tórax responde a la necesidad de desarrollar una solución tecnológica que permita mejorar la precisión y rapidez en la detección de esta enfermedad. El uso de la metodología CRISP-DM para el entrenamiento del modelo garantiza una estructura clara y sistemática para la recolección, preparación y análisis de los datos, permitiendo adaptar el proceso a las particularidades del problema clínico.
Fase 1. Comprensión del negocio
En el ámbito del diagnóstico clínico, la detección temprana y precisa de la tuberculosis es crucial para reducir su propagación y mejorar la tasa de recuperación. Sin embargo, el diagnóstico mediante radiografías de tórax presenta múltiples desafíos, incluyendo la disponibilidad limitada de especialistas capacitados, variaciones en la calidad de las imágenes, diferencias en los equipos utilizados y la subjetividad inherente a la interpretación visual, siendo limitaciones que a menudo derivan en retrasos o errores diagnósticos que afectan negativamente la eficacia de los tratamientos aplicados. Frente a este contexto, surge la necesidad de incorporar tecnologías automatizadas que complementen los métodos tradicionales, por lo que este estudio se enfoca en evaluar modelos de deep learning como TBNet, DenseNet121, VGG-16, ResNet50 y MobileNetV2, para automatizar la detección de tuberculosis a partir de imágenes radiográficas que mejoren la precisión diagnóstica en entornos con recursos limitados.
Fase 2. Comprensión de los datos
Se recopilaron los datos utilizados a partir de diferentes conjuntos de imágenes radiográficas disponibles en la plataforma Kaggle (Kiran y Jabeen, 2024; Rahman et al., 2020). El conjunto fue integrado, garantizando un equilibrio entre clases “Tuberculosis” y “Normal”, donde se aplicó un proceso de selección aleatoria para descartar imágenes de manera controlada. En la tabla 1, se observa la distribución del dataset unificado que contiene un total de 11,200 imágenes radiográficas de tórax distribuidas en: entrenamiento, validación y prueba.
Tabla 1.
Distribución por clases del conjunto de datos.
|
Clase |
Entrenamiento (80%) |
Validación (15%) |
Prueba (5%) |
|
Tuberculosis |
4,640 |
870 |
290 |
|
Normal |
4,640 |
870 |
290 |
Elaboración: Los autores.
La Figura 2 presenta ejemplos de las radiografías pulmonares extraídas del conjunto de datos de Kaggle, incluyendo imágenes de pacientes sin indicios de tuberculosis como radiográficas que evidencias signos o patrones asociados a la presencia y posible progresión de la enfermedad.
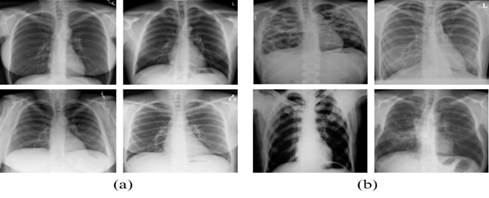
Figura 2. Radiografías de pulmón sin afectaciones (a); Radiografías de pulmón afectado por tuberculosis (b).
Fuente: Kiran y Jabeen (2024); Rahman et al. (2020)
Fase 3. Preparación de los datos
Se realizó un ajuste a todas las imágenes a una estándar de 224x224 píxeles, se aisló la región pulmonar y se aplicó una normalización dentro del rango [0, 1] para facilitar que la red neural convolucional pueda aprender los patrones más relevantes de cada imagen en el proceso de entrenamiento y validación como se puede observar en la tabla 2.
Tabla 2.
Técnicas de preprocesamiento aplicadas a las imágenes.
|
Transformación |
Aplicación |
Propósito |
|
Rotación |
±20° |
Permitir variabilidad en la orientación. |
|
Desplazamiento |
Horizontal y vertical (±20%) |
Simular diferentes posiciones. |
|
Zoom |
±20% |
Aumentar o reducir el tamaño. |
|
Ajuste de Brillo |
0.8 a 1.2 |
Variar la intensidad lumínica. |
|
Volteo Horizontal |
Aplicado de forma aleatoria |
Reflejar las imágenes. |
|
Inclinación |
±15° |
Simular deformaciones angulares. |
Elaboración: Los autores.
Se aplicó una serie de técnicas de aumento de datos para el entrenamiento de los modelos CNN para mejorar la capacidad del modelo para generalizar casos, reducir riesgos de sobreajuste y aumentar la robustez y precisión.
· Librerías y recursos utilizados
En este estudio, el preprocesamiento y la ampliación de datos se realizaron utilizando las bibliotecas TensorFlow y Keras. TenserFlow reconocida por su capacidad de construir modelos Deep Learning de forma eficiente con el enfoque de transformación automática de código en entrenamiento distribuido, apto para modelos de entornos heterogéneos (Sim et al., 2025). Por otro lado, Keras es una API de alto nivel que permite la construcción y entrenamiento de redes neuronales sobre TensorFlow y otros marcos de aprendizaje automático (Chicho y Sallow, 2021).
En cuanto al entrenamiento de los modelos, se realizó utilizando una computadora personal. Este entrenamiento se llevó a cabo localmente, aprovechando la capacidad de procesamiento de un procesador Intel® Core™ i9 y el soporte de la tarjeta gráfica NVIDIA GeForce RTX 4060, optimizando el desempeño del modelo mediante el uso de bibliotecas de deep learning como TensorFlow y Keras en Python.
Para evaluar el rendimiento del modelo de diagnóstico de tuberculosis, es fundamental comprender las siguientes métricas de clasificación:
![]() [1]
[1]
![]() [2]
[2]
![]() [3]
[3]
![]() [4]
[4]
Donde ![]() corresponde
a los valores obtenidos como verdaderos positivos,
corresponde
a los valores obtenidos como verdaderos positivos, ![]() al
valor obtenido como verdaderos negativos,
al
valor obtenido como verdaderos negativos, ![]() corresponde
a las predicciones catalogadas como falsos positivos y
corresponde
a las predicciones catalogadas como falsos positivos y ![]() que
corresponde a las predicciones catalogadas como falsos negativos.
que
corresponde a las predicciones catalogadas como falsos negativos.
![]() [5]
[5]
Fase 4. Modelado
Se evaluó cuatro arquitecturas de redes neuronales convolucionales como son: TBNet, DenseNet121, ResNet50 y MobileNetV2. Cada uno de estos modelos fue seleccionado en función de sus características específicas, que se ajustan a las necesidades del estudio.
· DenseNet121
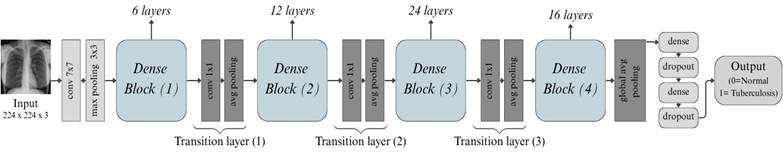
Figura 3. Arquitectura de DenseNet121.
Elaboración: Los autores.
Como se puede observar en la Figura 3, DenseNet121 es una red neuronal diseñada para reconocer características visuales complejas en imágenes. Su arquitectura se organiza en cuatro bloques llamados comúnmente Dense Blocks, que contienen 6, 12, 24 y 16 capas, respectivamente, donde cada bloque está compuesto por capas conectadas entre sí, lo que permite que cada capa aproveche las características aprendidas por las anteriores.
Entre los bloques, se utilizan capas de transición que reducen el tamaño de las imágenes, optimizando el modelo. Al final, se aplica una capa de Global Average Pooling (GAP), que convierte las características en un vector, seguido de una capa densa y una capa Dropout (40%) para evitar el sobreajuste. La salida se obtiene a través de una capa con activación sigmoide, que clasifica las imágenes como normales (0) o con tuberculosis (1). En el diagnóstico médico, DenseNet121 ha demostrado ser eficaz para detectar tuberculosis, logrando una precisión del 91%, una sensibilidad del 90% y una puntuación F1 del 89% (Madyono y Nabilah, 2025).
· MobileNetv2
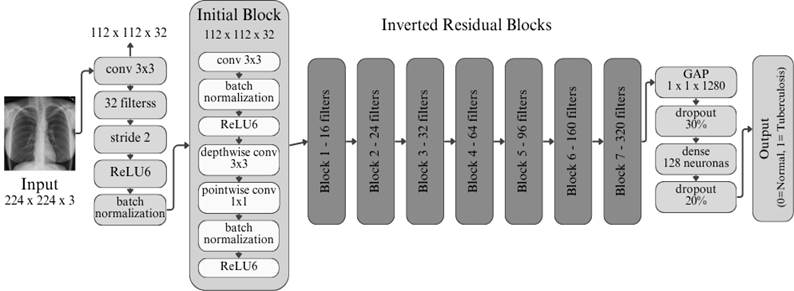
Figura 4. Arquitectura de MobileNetv2.
Elaboración: Los autores.
Por otro lado, como se muestra en la Figura 4, MobileNetv2 es una arquitectura optimizada para dispositivos móviles y entornos con recursos computacionales limitados, basada en bloques convolucionales invertidos con conexiones residuales lineales. Esta configuración permite reducir significativamente el número de parámetros y operaciones, manteniendo un desempeño competitivo (Hadi y Ali, 2021).
En la arquitectura, el modelo inicia con una capa de entrada de imágenes (224x224x3), seguida de una capa de convolución 3x3 con stride 2, que reduce las dimensiones. Esta primera etapa está acompañada de Batch Normalization y activación ReLU6, optimizando la extracción inicial de características, para posteriormente, organizarse en una serie de Bloques de Inversión Residual (Inverted Residual Blocks), que aprovechan convoluciones depthwise y pointwise para maximizar la eficiencia. El flujo culmina en una capa de Global Average Pooling, seguida de una capa densa de 128 neuronas (ReLU), una capa Dropout para regularización y una salida Sigmoide que permite la clasificación binaria (0 = Normal, 1 = Tuberculosis).
· ResNet50
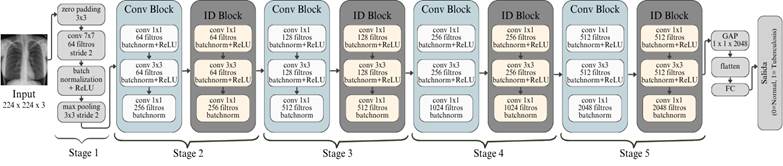
Figura 5. Arquitectura de ResNet50.
Elaboración: Los autores.
La Figura 5 presenta la arquitectura de ResNet50, una red profunda que se caracteriza por el uso de conexiones residuales que capturan características jerárquicas de las imágenes médicas.
Gracias a esta estructura, ResNet50 es capaz de extraer características altamente discriminativas, logrando un desempeño notable en el diagnóstico de enfermedades pulmonares, incluida la tuberculosis, lo que ha sido validado en estudios recientes que destacan su precisión y robustez en aplicaciones clínicas (Brun et y al., 2021).
· TBNet
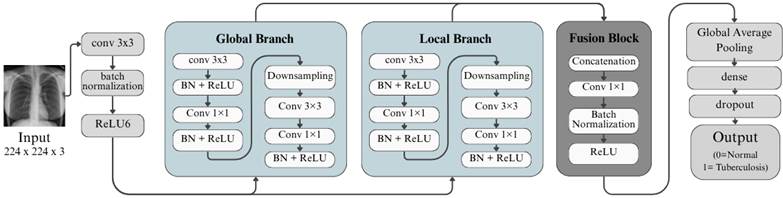
Figura 6. Arquitectura de TBNet.
Elaboración: Los autores.
Finalmente, en la Figura 6 se presenta TBNet, una arquitectura convolucional diseñada específicamente para la detección de tuberculosis en radiografías de tórax, caracterizada por su estructura de dos ramas paralelas.
El modelo inicia con una convolución 3×3 acompañada de normalización por lotes y activación ReLU, encargada de extraer las primeras características de la imagen. A partir de este punto, la red se divide en una rama global, que captura patrones amplios del campo pulmonar mediante bloques convolucionales profundos y operaciones de reducción espacial, y una rama local, que trabaja con convoluciones más ligeras para preservar detalles finos asociados a posibles lesiones tuberculosas.
Posteriormente, ambas ramas se integran en un bloque de fusión mediante concatenación y una convolución 1×1 que combina la información obtenida. La arquitectura continúa con una capa de Global Average Pooling, seguida de una capa densa y Dropout para reducir el sobreajuste, finalizando con una activación sigmoide que permite clasificar las radiografías como normales o con indicios de tuberculosis (Wong et al., 2022).
En la investigación, a partir del análisis, se utilizaron tres arquitecturas de redes neuronales convolucionales preentrenadas: DenseNet121, ResNet50, MobileNetV2 y TBNet, todas inicializadas con pesos en ImageNet. Esto permitió aprovechar el conocimiento adquirido por las redes durante su entrenamiento previo en un amplio conjunto de imágenes, maximizando su capacidad para reconocer patrones visuales complejos.
Durante la fase de transfer learning, las capas preentrenadas de cada modelo se mantuvieron congeladas, conservando el conocimiento adquirido en ImageNet. Solo las capas superiores personalizadas fueron entrenadas para adaptarse a la tarea específica de clasificación de tuberculosis. Posteriormente, en la fase de fine-tuning, se descongelaron las capas superiores de cada modelo.
Las imágenes utilizadas fueron redimensionadas a 224 × 224 píxeles, garantizando compatibilidad con las arquitecturas preentrenadas. El entrenamiento se realizó en lotes de 32 imágenes, utilizando el optimizador Adam, conocido por su capacidad para ajustar automáticamente las tasas de aprendizaje. La configuración incluyó una tasa de aprendizaje de 0.0001 durante la fase de Transfer Learning y 0.00001 en la fase de Fine-Tuning, optimizando la estabilidad del entrenamiento. En total, se entrenaron los modelos durante 40 épocas, distribuidas en un 60% para Transfer Learning y 40% para Fine-Tuning.
El rendimiento de los modelos fue monitoreado utilizando TensorBoard, una herramienta que permitió visualizar las curvas de pérdida y precisión tanto para los conjuntos de entrenamiento como de validación. Además, al finalizar el entrenamiento, se optimizó el umbral de clasificación utilizando el conjunto de validación, ajustando este umbral para maximizar la puntuación F1 y garantizar un equilibrio adecuado entre precisión y sensibilidad.
RESULTADOS
Evaluación de los modelos
Los resultados obtenidos para los modelos de redes neuronales convolucionales utilizados en este trabajo se presentan a continuación. Cada modelo fue entrenado y evaluado siguiendo una configuración uniforme de hiperparámetros, garantizando una comparación justa entre ellos. Las métricas clave de evaluación utilizadas para analizar su rendimiento fueron la precisión, el recall y el F1-Score, que permiten evaluar su capacidad para clasificar correctamente las imágenes en dos categorías: normales y con tuberculosis. Además, se generaron matrices de confusión para cada modelo, proporcionando una representación gráfica de sus predicciones.
La Figura 7 presenta las matrices de confusión obtenidas para los cuatro modelos evaluados (MobileNetV2, ResNet50, DenseNet121 y TBNet), lo que permite comparar de manera directa su capacidad para distinguir entre imágenes normales y casos de tuberculosis.
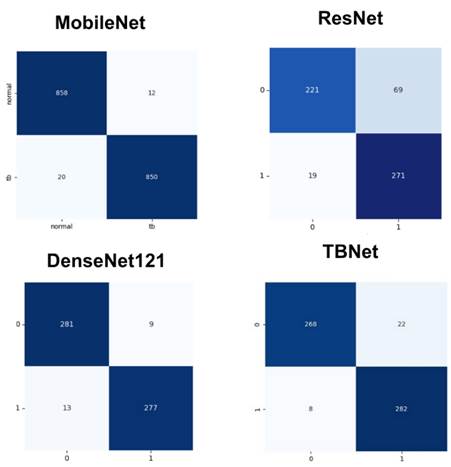
Figura 7. Matrices de confusión para todos los modelos.
Elaboración: Los autores.
En el caso de MobileNetV2, el modelo alcanza un desempeño consistente, con un elevado número de 850 verdaderos positivos y 858 verdaderos negativos, así como una cantidad moderada de 12 falsos positivos y 20 falsos negativos, evidenciando un comportamiento estable. Por su parte, ResNet50 muestra el rendimiento menos favorable, ya que presenta un mayor número de errores tanto en falsos positivos con 48 casos como en falsos negativos con 40 casos, lo que limita su precisión y su capacidad discriminativa frente a las demás arquitecturas. DenseNet121 exhibe una distribución de errores considerablemente más controlada, alcanzando 279 verdaderos negativos y 277 verdaderos positivos, con solo 11 falsos positivos y 13 falsos negativos. Este equilibrio refleja una arquitectura robusta y consistente en la clasificación binaria. Finalmente, TBNet demuestra el desempeño más sobresaliente del conjunto, especialmente en términos clínicos, al registrar únicamente 8 falsos negativos acompañado de 282 verdaderos positivos y 268 verdaderos negativos.
En conjunto, las matrices de confusión permiten identificar diferencias claras entre las arquitecturas. Mientras ResNet50 presenta una variabilidad elevada en sus errores, MobileNetV2 mantiene un rendimiento aceptable y DenseNet121 se distingue por su estabilidad general. No obstante, TBNet se posiciona como el modelo con el mejor comportamiento global, al combinar una alta tasa de aciertos con el número más bajo de falsos negativos, consolidándose como la alternativa más segura y efectiva para la detección de tuberculosis.
En la Figura 8 se evidencia diferencias claras en la estabilidad de las curvas de pérdida entre los modelos evaluados. DenseNet121 muestra la trayectoria más consistente, con una disminución progresiva y curvas de entrenamiento y validación muy próximas, lo que refleja un proceso de aprendizaje estable y sin fluctuaciones relevantes. ResNet50 presenta un patrón descendente aceptable, aunque con una convergencia más lenta. Por el contrario, MobileNetV2 y TBNet exhiben mayor variabilidad: MobileNetV2 presenta un pico abrupto de pérdida en validación, mientras que TBNet muestra oscilaciones repetidas a lo largo de las primeras épocas antes de estabilizarse. Estas fluctuaciones indican que, en términos puramente visuales.
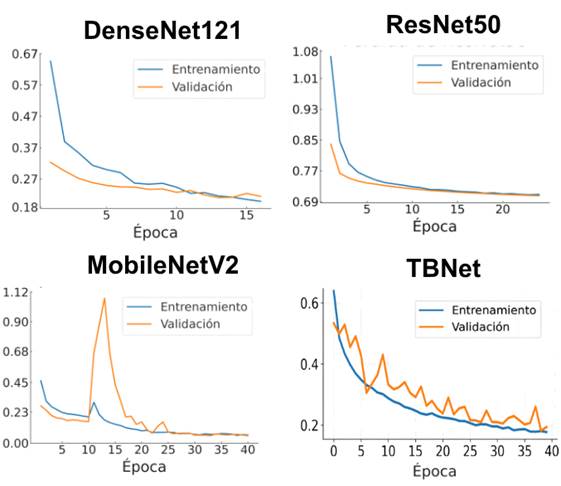
Figura 8. Curva de pérdida por época de los modelos evaluados.
Elaboración: Los autores.
Por otro lado, la Figura 9 presenta las curvas de precisión de entrenamiento y validación para los modelos DenseNet121, ResNet50, MobileNetV2 y TBNet, permitiendo comparar su comportamiento durante el proceso de aprendizaje. DenseNet121 muestra un incremento progresivo y estable de la precisión, con curvas cercanas y sin fluctuaciones bruscas, lo que indica una convergencia consistente. En contraste, ResNet50 exhibe una precisión más baja y considerablemente irregular, con oscilaciones marcadas tanto en entrenamiento como en validación, lo que evidencia dificultades para estabilizar el aprendizaje. MobileNetV2 alcanza valores altos de precisión, pero presenta una caída repentina en validación en las primeras épocas, seguida de una recuperación posterior, lo que refleja cierta inestabilidad inicial antes de converger. Por su parte, TBNet muestra una precisión que aumenta rápidamente y mantiene valores elevados a lo largo del entrenamiento, aunque con variaciones moderadas en la curva de validación que indican un proceso de ajuste más dinámico.
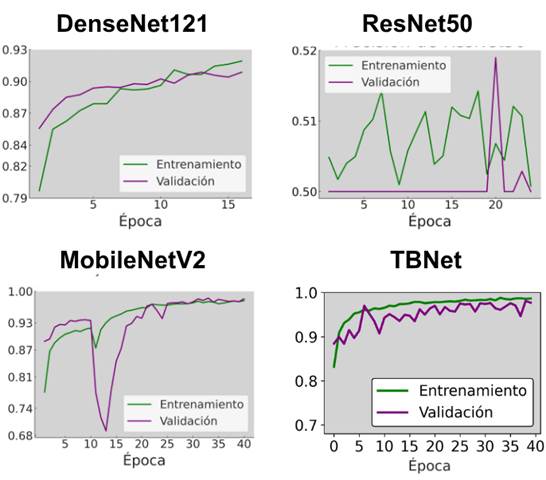
Figura 9. Curva de precisión por época de los modelos evaluados.
Elaboración: Los autores.
En conjunto, los resultados obtenidos permiten establecer una comparación sólida entre las cuatro arquitecturas evaluadas. DenseNet121 demostró ser el modelo con el entrenamiento más estable, presentando curvas de pérdida y precisión limpias, sin fluctuaciones bruscas y con una clara ausencia de señales de sobreajuste o subajuste. Sin embargo, al analizar el desempeño cuantitativo reflejado en las métricas finales se observa que TBNet supera consistentemente al resto de arquitecturas en las medidas más críticas para la detección de tuberculosis. En dichas medidas, TBNet alcanzó la mayor accuracy, precisión y un F1-score, situándose por encima del rendimiento logrado por MobileNetV2 y DenseNet121. Por ende, se determina que TBNet fue el modelo que presentó el mejor equilibrio entre aciertos y errores, destacándose especialmente por su precisión y su bajo número de falsos negativos, una característica esencial en aplicaciones clínicas donde la omisión de un caso positivo podría tener consecuencias críticas.
A partir del análisis, en la tabla 3 se muestra las métricas obtenidas en la evaluación de los modelos CNN.
Tabla 3.
Métricas obtenidas en la evaluación de los modelos CNN.
|
Métricas |
ResNet50 |
DenseNet121 |
MobileNetV2 |
TBNet |
|
Accuracy |
84,83% |
96,21% |
98,16% |
99,14% |
|
Precision |
79,71% |
96,85% |
98,93% |
99.52% |
|
Recall |
93,45% |
95,52% |
98,93% |
98,28% |
|
F1-Score |
86,03% |
96,18% |
98,93% |
99,13% |
Elaboración: Los autores.
DISCUSIÓN
Los resultados del estudio confirman que el uso de arquitecturas de redes neuronales convolucionales es una alternativa confiable para el apoyo del diagnóstico automatizado de la tuberculosis a partir de radiografías de tórax. El desempeño superior de TBNet frente a arquitecturas consolidadas como DenseNet121, ResNet50 y MobileNetV2 evidencia que los modelos diseñados para tareas clínicas pueden superar a modelos genéricos preentrenados, incluso cuando estos presentan procesos de entrenamiento más estables. Este hallazgo coincide con lo señalado por Lu et al. (2022) y Wong et al. (2022), quienes destacan que las arquitecturas especializadas logran capturar patrones patológicos más relevantes al incorporar mecanismos de atención contextual y análisis multiescala.
Si bien DenseNet121 mostró una mayor estabilidad durante el entrenamiento, reflejada en curvas de pérdida y precisión más uniformes, los resultados cuantitativos demuestran que la estabilidad visual del aprendizaje no siempre se traduce en un mejor rendimiento clínico. En este sentido, TBNet logró maximizar métricas críticas como accuracy, precisión y F1-Score, además de minimizar los falsos negativos, indicador clave en escenarios diagnósticos. Esta observación es consistente con estudios recientes que advierten que, en aplicaciones médicas, la priorización de métricas clínicas debe prevalecer sobre indicadores computacionales, ya que el impacto de un falso negativo puede comprometer el tratamiento oportuno del paciente (Rahman et al., 2020; Sánchez Figueroa et al., 2024).
El bajo desempeño relativo de ResNet50 en este estudio ha sido reportado en investigaciones similares, donde arquitecturas profundas sin mecanismos específicos de atención o fusión de características tienden a presentar dificultades para discriminar lesiones sutiles en radiografías pulmonares (Brun et al., 2021; Kotei y Thirunavukarasu, 2024). Por el contrario, MobileNetV2 demostró un rendimiento competitivo, lo que confirma su utilidad en entornos con recursos computacionales limitados. Sin embargo, su menor capacidad para reducir falsos negativos lo sitúa por debajo de TBNet cuando se considera su aplicabilidad clínica directa.
La arquitectura dual de TBNet, basada en la integración de información global y local, permitió una representación más rica de las estructuras pulmonares y de las lesiones asociadas a la tuberculosis. Este enfoque coincide con tendencias actuales en deep learning médico, donde la combinación de múltiples escalas y contextos anatómicos mejora significativamente la capacidad discriminativa de los modelos (Lu et al., 2022; Wong et al., 2022). En este sentido, los resultados obtenidos refuerzan la hipótesis de que los modelos diseñados específicamente para tareas médicas, más que las arquitecturas generalistas, representan una línea de desarrollo prioritaria para sistemas de apoyo al diagnóstico.
Finalmente, el uso de la metodología CRISP-DM permitió estructurar de manera rigurosa el proceso de desarrollo, entrenamiento y evaluación de los modelos, asegurando la reproducibilidad y validez de los resultados. Tal como señalan Schröer et al. (2021) y Tamayo y Sauñe (2024), la aplicación de metodologías formales en proyectos de machine learning en salud, no solo mejora la calidad técnica del modelo, sino que también facilita su futura integración en entornos clínicos reales. En conjunto, los hallazgos de este estudio consolidan a TBNet como una herramienta prometedora para el apoyo al diagnóstico temprano de la tuberculosis, especialmente en contextos con limitaciones de recursos y alta carga epidemiológica.
CONCLUSIONES
Aunque DenseNet121 presentó la curva de pérdida más estable y un proceso de aprendizaje visualmente más consistente, los resultados cuantitativos mostraron que la estabilidad gráfica no siempre se traduce en el mejor desempeño final. En este estudio, TBNet demostró ser el modelo más eficiente para la detección de tuberculosis, aun cuando su curva de pérdida exhibió oscilaciones notables durante las primeras épocas. Estas variaciones no afectaron su capacidad discriminativa y el modelo logró converger adecuadamente y produjo las métricas más altas en precisión, accuracy y F1-Score.
Las matrices de confusión brindaron el argumento más sólido para su selección, donde TBNet obtuvo el menor número de falsos negativos, considerada una métrica crítica en el diagnóstico clínico donde no identificar un caso positivo puede comprometer el tratamiento y la contención de la enfermedad. La arquitectura dual de TBNet, basada en dos ramas paralelas que capturan simultáneamente características globales y detalles locales, permitió una extracción de características más rica que las arquitecturas convencionales evaluadas.
En conjunto, los resultados técnicos confirman que la superioridad de TBNet no deriva de la forma de su curva de pérdida, sino de su desempeño final en las métricas más relevantes para la práctica clínica. Por ello, TBNet se posiciona como la opción más adecuada para apoyar el diagnóstico automatizado de tuberculosis, priorizando la reducción de errores críticos y la detección oportuna de casos positivos.
CONFLICTO DE INTERÉS
Los autores declaran que no tienen conflicto de interés en la publicación de este artículo.
FINANCIAMIENTO
Trabajo financiado por los autores.
AGRADECIMIENTO
Expresamos nuestro sincero agradecimiento a nuestras familias por su apoyo incondicional durante todo este proceso. De manera especial, extiendo mi gratitud a Madelayne Cando, por su apoyo y compañía durante el desarrollo académico y personal en este proceso, y a Darío Arias, por su constante apoyo incondicional y presencia en esta etapa.
REFERENCIAS CONSULTADAS
Brun, A. L., Chabi, M. L., Picard, C., Mellot, F., y Grenier, P. A. (2021). Lung Transplantation: CT Assessment of Chronic Lung Allograft Dysfunction (CLAD). Diagnostics, 11(5), 817. https://doi.org/10.3390/diagnostics11050817
Chicho, B. T., y Sallow, A. B. (2021). A Comprehensive Survey of Deep Learning Models Based on Keras Framework. Journal of Soft Computing and Data Mining, 2(2), 49-62. https://doi.org/10.30880/JSCDM.2021.02.02.005
Hadi, M. A., y Ali, H. I. (2021). Control of COVID-19 system using a novel nonlinear robust control algorithm. Biomedical Signal Processing and Control, 64, 102317. https://doi.org/10.1016/j.bspc.2020.102317
Kiran, S., y Jabeen, I. (2024). Tuberculosis Chest X-rays Images. Kaggle. https://doi.org/10.34740/KAGGLE/DSV/10304386
Kotei, E., y Thirunavukarasu, R. (2024). Tuberculosis Detection From Chest X-Ray Image Modalities Based on Transformer and Convolutional Neural Network. IEEE Access, 12, 97417–97427. https://doi.org/10.1109/ACCESS.2024.3428446
Liu, Y. E., Mabene, Y., Camelo, S., Rueda, Z. V., Pelissari, D. M., Dockhorn Costa Johansen, F., Huaman, M. A., Avalos-Cruz, T., Alarcón, V. A., Ladutke, L. M., Bergman, M., Cohen, T., Goldhaber-Fiebert, J. D., Croda, J., y Andrews, J. R. (2024). Mass incarceration as a driver of the tuberculosis epidemic in Latin America and projected effects of policy alternatives: a mathematical modelling study. The Lancet Public Health, 9(11), e841–e851. https://doi.org/10.1016/S2468-2667(24)00192-0
Lu, S. Y., Wang, S. H., Zhang, X., y Zhang, Y. D. (2022). TBNet: a context-aware graph network for tuberculosis diagnosis. Computer Methods and Programs in Biomedicine, 214, 106587. https://doi.org/10.1016/J.CMPB.2021.106587
Madyono, M., y Nabilah, A. (2025). Detection of Tuberculosis Disease in Lung X-ray Images Using the DenseNet121 Method. International Journal of Engineering, Science and Information Technology, 5(2), 379–385. https://doi.org/10.52088/ijesty.v5i2.853
Rahman, T., Khandakar, A., Kadir, M. A., Islam, K. R., Islam, K. F., Mazhar, R., Hamid, T., Islam, M. T., Kashem, S., Mahbub, Z. Bin, Ayari, M. A., y Chowdhury, M. E. H. (2020a). Reliable Tuberculosis Detection Using Chest X-Ray With Deep Learning, Segmentation and Visualization. IEEE Access, 8, 191586–191601. https://doi.org/10.1109/ACCESS.2020.3031384
Romero García, C. V., Saraguro Reyes, C. M., Mazon Olivo, B. E., y Morocho Román, R. F. (2025). Agricultura de precisión en la producción de banano. Revisión sistemática. Ingenium et Potentia, 7(12), 50–76. https://doi.org/10.35381/I.P.V7I12.4450
Sánchez Figueroa, L. S., Guillén Muñóz, V. A., Pérez Pérez, J. D., Rodríguez Abrego, P. A., Caprile Mata, C. M., y Cartagena López, K. L. (2024). Actualización en la detección de la tuberculosis a través de pruebas moleculares. Alerta, Revista científica del Instituto Nacional de Salud, 7(2), 184–190. https://doi.org/10.5377/alerta.v7i2.17129
Schröer, C., Kruse, F., y Gómez, J. M. (2021). A systematic literature review on applying CRISP-DM process model. Procedia Computer Science, 181, 526–534. https://doi.org/10.1016/J.PROCS.2021.01.199
Sim, Y., Shin, W., y Lee, S. (2025). Automated code transformation for distributed training of TensorFlow deep learning models. Science of Computer Programming, 242, 103260. https://doi.org/10.1016/J.SCICO.2024.103260
Tamayo, J. J., y Sauñe, A. B. (2024). Soluciones de TI basadas en machine learning aplicables a problemas del sistema de salud peruano. Revista Científica: BIOTECH AND ENGINEERING, 4(1), 156–163. https://doi.org/10.52248/EB.VOL4ISS1.116
Wong, A., Lee, J. R. H., Rahmat-Khah, H., Sabri, A., Alaref, A., y Liu, H. (2022). TB-Net: A Tailored, Self-Attention Deep Convolutional Neural Network Design for Detection of Tuberculosis Cases From Chest X-Ray Images. Frontiers in Artificial Intelligence, 5, 827299. https://doi.org/10.3389/FRAI.2022.827299
©2026 por los autores. Este artículo es de acceso abierto y distribuido según los términos y condiciones de la licencia Creative Commons Atribución-NoComercial-CompartirIgual 4.0 Internacional (CC BY-NC-SA 4.0) (https://creativecommons.org/licenses/by-nc-sa/4.0/).